特点:
•可以检测基因组DNA样品的碱基位点数量
•采用方便的比色法检测
•检测范围:40个碱基位点/ 1×105 碱基对DNA

活动进行中
订购满5000元,200元礼品等你拿
DNA损伤丨从实验思路到检测指标 PDF下载
| 指标 | 关联指标干货参考(点击查看) | 检测指标(点击查看) |
| DNA损伤检测 |
γ-H2AX | DNA损伤检测试剂盒- γH2AX(绿、红、深红) |
| AP位点 | DNA损伤检测试剂盒(AP位点法) | |
| 核小体 | Nucleolus Bright (绿、红) | |
| 细胞周期 | Cell Cycle Assay Kit – PI/RNase Staining | |
| DNA损伤关联指标 | 氧化损伤 | DMPO |
| BMPO | ||
| TEMP | ||
| SOD Assay Kit | ||
| DPPH Antioxidant Assay Kit | ||
| 凋亡 | Annexin V, FITC Apoptosis Detection Kit/PI | |
| Annexin V, 633 Apoptosis Detection Kit/PI | ||
| Cell Cycle Assay Solution Deep Red/Blue | ||
| JC-1 MitoMP Detection Kit | ||
| Caspase-3 Assay Kit | ||
| 铁死亡 | Lipid Peroxidation Probe -BDP 581/591 C11 | |
| Liperfluo | ||
| GSSG/GSH Quantification Kit II | ||
| Iron Assay Kit | ||
| Mito-FerroGreen | ||
| 衰老 | Cellular Senescence Detection Kit – SPiDER-βGal | |
| Cellular Senescence Plate Assay Kit – SPiDER-βGal | ||
| SPiDER-βGal | ||
| 自噬 | Mitophagy Detection Kit | |
| DALGreen – Autophagy Detection | ||
| DAPGreen – Autophagy Detection | ||
| DAPRed – Autophagy Detection |
*点击即可跳转至详情页
试剂盒内含

产品概述
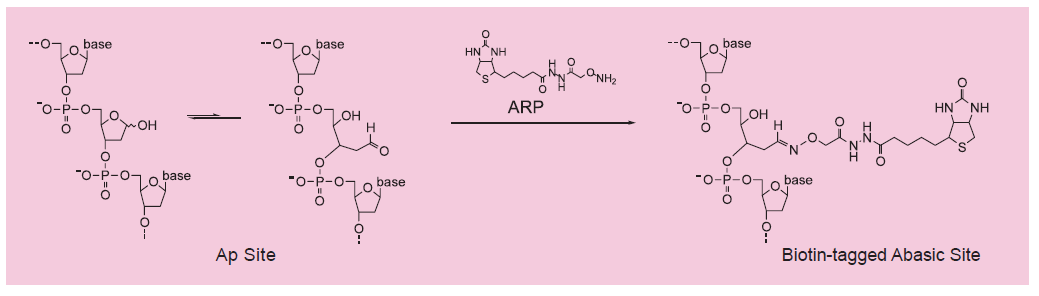
DNA的氧化损伤是由于DNA与活性氧 (ROS) 尤其是羟自由基之间的相互作用造成的。由超氧阴离子和过氧化氢通过Fenton反应产生的羟自由基在DNA中产生多重修饰。羟自由基对脱氧核糖基团的氧化攻击将导致DNA释放自由碱基,产生链断裂、各种糖修饰以及单个无碱基位点 (AP位点) 。事实上AP位点是由ROS产生的损伤的主要类型。醛反应性探针 (ARP;N’-氨氧基甲基羰基肼-D-生物素) 特异性地与AP位点的开环上的醛基反应。通过这个反应可以检测到导致醛基形成的DNA修饰。用过量ARP试剂反应后,DNA上的所有AP位点均用生物素做了标签。可以用亲和素-生物素法,用连接到亲和素上的过氧化物酶或碱性磷酸酶做比色法检测来对这些带有生物素标签的AP位点进行计数。DNA损伤定量试剂盒包含用于检测每1×105个碱基对中1-40个AP位点的所有必需溶液。
*本试剂盒仅适用于基因组DNA中无碱基位点 (Abasic Sites)的检测。
原理
DNA损伤在除了复制过程中的DNA聚合酶错误外,保留有机体遗传信息的DNA在体内还受到环境辐射,紫外线,化学物质(如烷基化剂)和代谢物(如活性氧)的破坏,接收。如果这些错误和伤害得不到适当的修复,它们将导致突变,从而导致癌症和衰老。
DNA损伤部位有修复结构,其中之一就是去除碱修复。此时,将出现一个称为AP位点的基础位点(apurinic/apyrimidinic位点)。换句话说,AP位点的检测是可以测量DNA损伤位点的有效方法。
ARP(N’-氨基氧基甲基羰基肼基-D-生物素)是已知可以与该AP位点进行生物素特异结合的试剂。-Nucleostain-DNA Damage Quantification Kit-AP Site Counting-是一种可以通过使用ARP将DNA生物素化并固定在96孔微板上,可以简单地定量样品DNA中的AP 位点的试剂盒。
该试剂盒包含具有与AP位点的标准DNA,并且可以使用HRP标记的链霉亲和素通过生物素检测方法对AP位点进行定量。
使用方法请看实验例。
*冷藏保存中或恢复到室温时,有时会在管状管中看到水滴状的液粒。
这是由干燥防止剂液粒化而成的,产品的性能没有问题。
*本套装中含有加入了溶液的试剂组件。
溶液可能会粘附在试管或瓶盖的内壁上,因此在打开前应先摇匀
技术信息
除试剂盒以外必要的物品
・10μl,200μl,1 ml微量移液器(可变型)
・200μl多通道移液器(可变型)
・培养箱(37℃)
・酶标仪
・0.5 ml,1.5 ml离心管
・离心机
・DNA纯化试剂盒
・纸巾
本公司出售各种DNA纯化试剂盒。
Get pureDNA Kit-Cell, Tissue(Code:GK03)
关于DNA提纯的问题,请咨询同仁化学。
操作方法

常见问题Q&A
| Q1: 是否可以创建40个以上AP位点/ 100,000个标准溶液? |
| A1:该试剂盒的标准DNA是0-40个AP位点/ 100,000ARP-DNA。但我们已经准备了多达50个AP站点/ 100,000个的ARP-DNA(暂时没有做到更多)。从理论上讲,可以制备更多的ARP-DNA,但这么做的话我们无法保证和保持校准线的线性程度。
如果要检测更高浓度的碱基损伤,但样品DNA没有相同浓度的损伤,您可以用0AP 位点-DNA(测定范围内稀释)后再进行测定,之后再换算实际AP数比较好。 |
| Q2: AP位点是DNA的一个基本损伤位点,被用作氧化应激的指标,除了AP位点之外,还有其他氧化应激指标吗? |
| A2:我们创建了一个文档,概述了氧化应激的各种标记物。
从实验者的角度,描述了每种氧化应激标记的特性和检测样品的处理方法,因此请参考以下文档(日文)。 https://www.dojindo.co.jp/technical/beginner/stressmarker.pdf |
| Q3:建议在DNA的纯度为吸光度为1.8以上进行检测。是否能够测量至1.5? |
| A3:因为吸光度在1.5内没有做过类似实验,所以暂时无法确认。
我们用吸光度为1.6~1.7的DNA进行检测,但不能看到很大的变化。 所以推荐1.8或更高。 纯度低的话可能意味着蛋白质污染。低纯度蛋白质具有阻断作用,可能使DNA粘附在板上。 请使用尽可能高的纯度,因为它可能会阻止这种情况 |
| Q4:是否可以存储提取的DNA而无需将其转换为ARP? |
| A4:存储是不可取的,因为这会增加AP位点的数量。 (冷藏和冷冻)
如果要在不进行ARP化的情况下进行存储,请使用乙醇沉淀,制粒并冷冻保存。AP站点确实是容易发生剪切的部位。 但是,如果以catallist free的状态保存的话,即使在3’侧有断开也不会影响ARP的检测。 相反,如果提取的DNA储存在非冷冻状态的环境中, 由自然产生的去除碱形成的AP位点是一个更重要的问题。 所以在这种情况下,如果你的样品在正确存储标准DNA是可以进行修饰的。 ARP修饰后AP位点稳定,建议提取后迅速进行APR处理。 |
| Q5:这个试剂盒可以检测什么? |
| A5:您可以定量DNA中的AP位点。AP位点是[apurinic / appyrimidinic site]的缩写,作用于受损的DNA。一般在去除碱修复过程中会出现。DNA的损伤除了复制时DNA polymerase的错误之外,还受到紫外线,化学物质(如烷基化剂)和代谢物(如活性氧)的影响。
通过测定AP位点可以检测DNA损伤。 |
| Q6:这个试剂盒能测定的样品数量是多少? |
| A6:20samples为20个样品。
每个Filtration Tube都有20个samples。 因为一个样品一般使用一个Filtration Tube,所以这个Tube的数量即是可测定的样品数。 20samples的测定用的板也是1块板。 |
| Q7:96孔板和过滤管中有溶液剩余,请告诉我废弃的方法 |
| A7:多余的溶液请在确认以下成分信息后,按照政策规定的废弃规则废弃。
<配套试剂成分> ・ARP-DNA standard solution(稀释水后废弃,不含有害成分) ・ARP solution(稀释后作废,不含有害成分) ・DNA binding solution(稀释水后废弃,不含有害成分) ・Washing buffer(稀释水后废弃,不含有害成分) ・HRP-Striptavidin(稀释水后废弃,不含有害成分) ・TE buffer(可溶于水,EDA・2NA 0.1%以下) ・Substrate solution(稀释水后废弃,不含有害成分) |
参考文献
1) A. Sancar and G. B. Sancar, “DNA Repair Enzymes”, Annu. Rev. Biochem., 1988, 57, 29.
2) T. Lindahl and B. Nyberg, “Rate of Depurination of Native Deoxyribonucleic Acid”, Biochemistry, 1972, 11, 3610.
3) M. Liuzzi and M. Talpaert-Borle, “A New Approach to the Study of the Base-excision Repair Pathway Using Methoxyamine”, J. Biol. Chem., 1985, 260, 5252.
4) M. Weinfeld, M. Liuzzi and M. C. Paterson, “Response of Phage T4 Polynucleotide Kinase Toward Dinucleotides Containing Apurinic Sites: Design of a 32P-postlabeling Assay for Apurinic Sites in DNA”, Biochemistry, 1990, 29, 1737.
5) B. X. Chen, K. Kubo, H. Ide, B. F. Erlanger, S. S. Wallace and Y. W. Kow, “Properties of a Monoclonal Antibody for the Detection of Abasic Sites, a Common DNA Lesion”, Mutat. Res., 1992, 273, 253.
6) J. A. Gralnick and D. M. Downs, “The YggX Protein of Salmonella enterica Is Involoved in Fe(II) Trafficking and Minimizes the DNA Damage Cause by Hydroxyl Radicals:Residue CYS-7 is Essential for YggX Function”, J. Biol. Chem., 2003, 278, 20708.
7) J. W. Pippin, R. Durvasula, A. Petermann, K. Hiromura, W. G. Couser and S. J. Shankland, “DNA Damage is a Novel Response to Sublytic Complement C5b-9 Induced Injury in Podocytes”, J. Clin. Invest., 2003, 111, 877.
8) S. Watanabe, T. Ichimura, N. Fujita, S. Tsuruzono, I. Ohki, M. Shirakawa, M. Kawasuji and M. Nakao, “Methylated DNA-binding Domain 1 and Methylpurine DNA Glycosylase Link Transcriptional Repression and DNA Repair in Chromatin”, Proc. Natl. Acad. Sci. USA, 2003, 100, 12859.
9) M. Endres, M. Ahmadi, I. Kruman, D. Biniszkiewicz, A. Meisel and K. Gertz, “Folate Deficiency Increases Postischemic Brain Injury”, Stroke, 2005, 36, 321.
10) J.-M. Li, M. Mogi, K. Tsukuda, H. Tomochika, J. Iwanami, L.-J. Min, C. Nahmias, M. Iwai and M. Horiuchi, “Angiotensin II-Induced Neural Differentiation via Angiotensin II Type 2 (AT2) Receptor-MMS2 Cascade Involving Interaction between AT2 Receptor-Interacting Protein and Src Homology 2 Domain-Containing Protein-Tyrosine Phosphatase 1”, Mol. Endocrinolo., 2007, 21(2):499.
11) D. R. McNeill and D. M. Wilson III, “A Dominant-Negative Form of the Major Human Abasic Endonuclease Enhances Cellular Sensitivity to Laboratory and Clinical DNA-Damaging Agents”, Mol. Cancer Res., 2007, 5(1), 61.
